REDES CON ESTRUCTURA FUNCIONAL
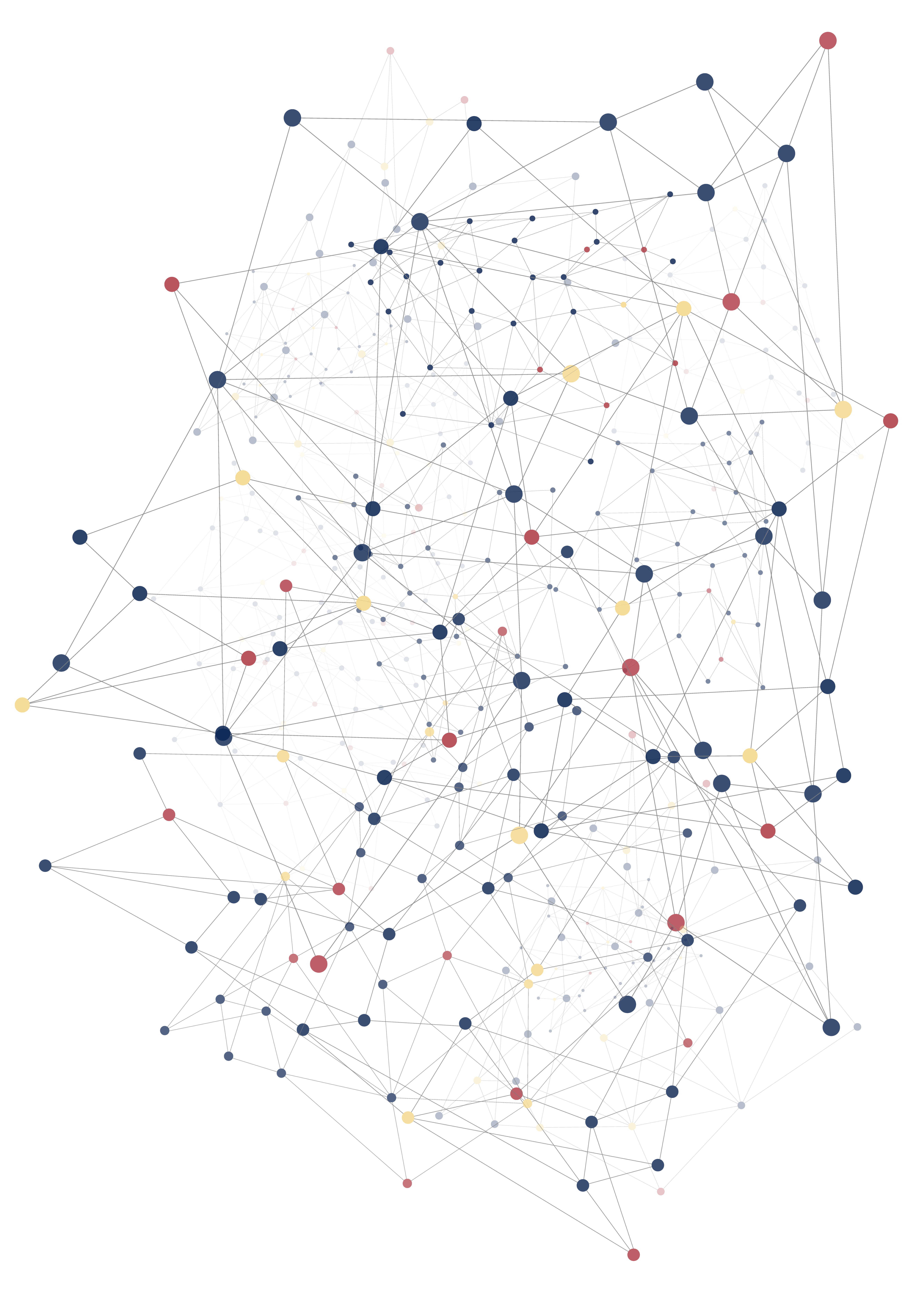
Mediante modelos Bayesianos construimos una red usando los datos de expresión de líneas celulares o muestras clínicas. La principal característica de este tipo de redes es que poseen estructura funcional, es decir, es posible establecer nodos con una función biológica mayoritariamente representada. De esta manera es posible caracterizar grupos de pacientes a nivel funcional, pudiendo estudiar las diferencias existentes en los procesos biológicos.

Publicaciones Relacionadas

1 junio, 2015
Cancer Research
Combined Label-Free Quantitative Proteomics and microRNA Expression Analysis of Breast Cancer Unravel Molecular Differences with Clinical Implications.
Gámez-Pozo et al.

15 abril, 2020
BMC Cancer
Computational models applied to metabolomics data hints at the relevance of glutamine metabolism in breast cancer.
Trilla-Fuertes et al.

8 enero, 2018
Oncotarget
Molecular characterization of breast cancer cell response to metabolic drugs.
Trilla-Fuertes et al.

7 febrero, 2019
scientific reports
A novel approach to triple-negative breast cancer molecular classification reveals a luminal immune-positive subgroup with good prognoses.
Prado-Vázquez et al.

12 junio, 2018
Oncotarget
Probabilistic graphical models relate immune status with response to neoadjuvant chemotherapy in breast cancer.
Zapater-Moros et al.

17 noviembre, 2017
scientific reports
Urothelial cancer proteomics provides both prognostic and functional information.
De Velasco et al

10 mayo, 2019
scientific reports
Melanoma proteomics suggests functional differences related to mutational status.
Trilla-Fuertes et al.

19 abril, 2020
Molecular & Cellular Proteomics
Genetic Profile and Functional Proteomics of Anal Squamous Cell Carcinoma: Proposal for a Molecular Classification.
Trilla-Fuertes et al.
Algoritmos clasificatorios
Análisis de capas de información biológica
El análisis de la información biológica entendiéndola como un conjunto de informaciones complementarias pero independientes, nos permite profundizar en algunos aspectos claves, como puede ser la relación del sistema inmune del paciente con el tumor. La combinación del “sparse k-means” y el “consensus clustering algorithm” permiten definir diferentes capas de información en un conjunto de datos. De esta manera es posible separar la información en función de su importancia en la clasificación de las muestras.
Publicaciones Relacionadas

28 junio, 2019
BMC Cancer
Biological molecular layer classification of muscle-invasive bladder cancer opens new treatment opportunities.
Trilla-Fuertes et al.

7 febrero, 2019
scientific reports
A novel approach to triple-negative breast cancer molecular classification reveals a luminal immune-positive subgroup with good prognoses.
Prado-Vázquez et al.

17 noviembre, 2017
scientific reports
Urothelial cancer proteomics provides both prognostic and functional information.
De Velasco et al.
Modelización computacional
del metabolismo
El “Flux Balance Analysis” es una técnica que permite modelar de manera computacional una red metabólica. Se utilizaba tradicionalmente en microbiología, pero con la aparición de reconstrucciones cada vez más completas del metabolismo humano se ha ampliado su aplicación a campos como el cáncer. Sirve para caracterizar diferencias a nivel de rutas metabólicas y para predecir la tasa de crecimiento de un microorganismo o de un tumor.
Publicaciones Relacionadas

30 agosto, 2017
scientific reports
Functional proteomics outlines the complexity of breast cancer molecular subtypes.
Gámez-Pozo et al.

8 junio, 2018
Oncotarget
Molecular characterization of breast cancer cell response to metabolic drugs.
Trilla-Fuertes et al.

28 junio, 2019
BMC Cancer
Biological molecular layer classification of muscle-invasive bladder cancer opens new treatment opportunities.
Trilla-Fuertes et al.

10 mayo, 2019
scientific reports
Melanoma proteomics suggests functional differences related to mutational status.
Trilla-Fuertes et al.
Elaboración de predictores
Los predictores de clase permiten elaborar un predictor que clasifique las muestras clínicas en diferentes grupos. Por otro lado, los predictores de supervivencia permiten clasificar las muestras según alto o bajo riesgo de recaída. Estos predictores se definen a partir de datos de expresión de genes, proteínas, metabolitos o miRNAs. También es posible elaborar predictores a partir de los datos obtenidos en las redes Bayesianas o los modelos metabólicos.

Publicaciones Relacionadas

8 junio, 2017
PLOS ONE
Prediction of adjuvant chemotherapy response in triple negative breast cancer with discovery and targeted proteomics.
Gámez-Pozo et al.

17 noviembre, 2017
scientific reports
Urothelial cancer proteomics provides both prognostic and functional information.
De Velasco et al.

3 octubre, 2019
Future Oncology
Computational metabolism modeling predicts risk of distant relapse-free survival in breast cancer patients.
Trilla-Fuertes et al.

30 agosto, 2017
scientific reports
Functional proteomics outlines the complexity of breast cancer molecular subtypes.
Gámez-Pozo et al.
Análisis de patrones de expresión diferenciales
Mediante técnicas como el “Significance Analysis of Microarrays” (SAM) podemos establecer diferencias en los patrones de expresión de genes, proteínas, miRNAs o metabolitos entre dos o más grupos de pacientes. Esta herramienta ha demostrado ser útil, por ejemplo, en la definición de grupos moleculares de novo. El SAM nos proporciona una lista de genes diferenciales entre los grupos.
Publicaciones Relacionadas

30 agosto, 2017
scientific reports
Functional proteomics outlines the complexity of breast cancer molecular subtypes.
Gámez-Pozo et al.

28 febrero, 2020
PLOS ONE
miRNA profiling in renal carcinoma suggest the existence of a group of pro-angionenic tumors in localized clear cell renal carcinoma.
Trilla-Fuertes et al.

19 abril, 2020
Molecular & Cellular Proteomics
Genetic Profile and Functional Proteomics of Anal Squamous Cell Carcinoma: Proposal for a Molecular Classification.
Trilla-Fuertes et al.

10 mayo, 2019
scientific reports
Melanoma proteomics suggests functional differences related to mutational status.
Trilla-Fuertes et al.

1 junio, 2015
Cancer Research
Combined Label-Free Quantitative Proteomics and microRNA Expression Analysis of Breast Cancer Unravel Molecular Differences with Clinical Implications.
Gámez-Pozo et al.
Redes dirigidas
Mediante la elaboración de redes Bayesianas basadas en gráficos dirigidos es posible caracterizar las relaciones existentes entre proteínas o genes de manera jerarquizada. Esta aproximación permite establecer hipótesis acerca de redes de regulación sin necesidad de conocimiento previo.

Publicaciones Relacionadas

11 junio, 2020
PLOS ONE
Bayesian networks established functional differences between breast cancer subtypes.
Trilla-Fuertes et al.


